Công Nghệ Sinh Học
Một cách thiết kế mồi cho phản ứng PCR
- Chi tiết bài viết
- Bài viết liên quan
RT-qPCR là một trong những kỹ thuật quan trọng và phổ biến trong các phòng thí nghiệm sinh học phân tử. RT-qPCR giúp định lượng sự có mặt của một gene một cách nhanh chóng từ sản phẩm của phản ứng cDNA. Việc thiết kế mồi (primer design) cho RT-qPCR có thể được thực hiện dễ dàng bằng các công cụ như Primer BLAST hay Primer3Plus. Trong bài viết này, IBSG xin giới thiệu đến bạn đọc một phương pháp thiết kế mồi đơn giản và nhanh chóng bằng trình duyệt genome (genome browser) của UCSC và công cụ PrimerQuest của IDT.
Để minh họa cho việc thiết kế mồi, bài viết lấy gene Tlr7 của chuột Mus musculus làm thí dụ.
Bước 1: Xác định các exon để thiết kế
Một gene có thể có nhiều biến thể (transcript variant), do đó cặp mồi phải được thiết kế sao cho có thể xác định được tất cả các transcript variant này. Đầu tiên, các bạn truy cập UCSC Genome Browser tại địa chỉ: https://genome.ucsc.edu/index.html.
Trong mục Our Tool, bạn chọn Genome Browser. Mục Browse/Select Species, bạn chọn Mouse. Mục Mouse Assembly, bạn chắc chắn rằng Dec. 2011 (GRCm38/mm10) được chọn.
Mục Position/Search Term, bạn điền tên gene Tlr7. Trình duyệt sẽ hiện ra gợi ý gene ngay sau khi bạn điền. Click chọn một gene phù hợp với gene mà bạn quan tâm. Trong trường hợp này là Tlr7 (Mus musculus toll-like receptor 7 (Tlr7) transcript variant 3 mRNA from RefSeq NM_133211).
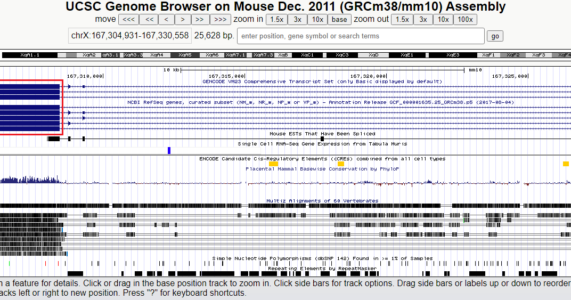
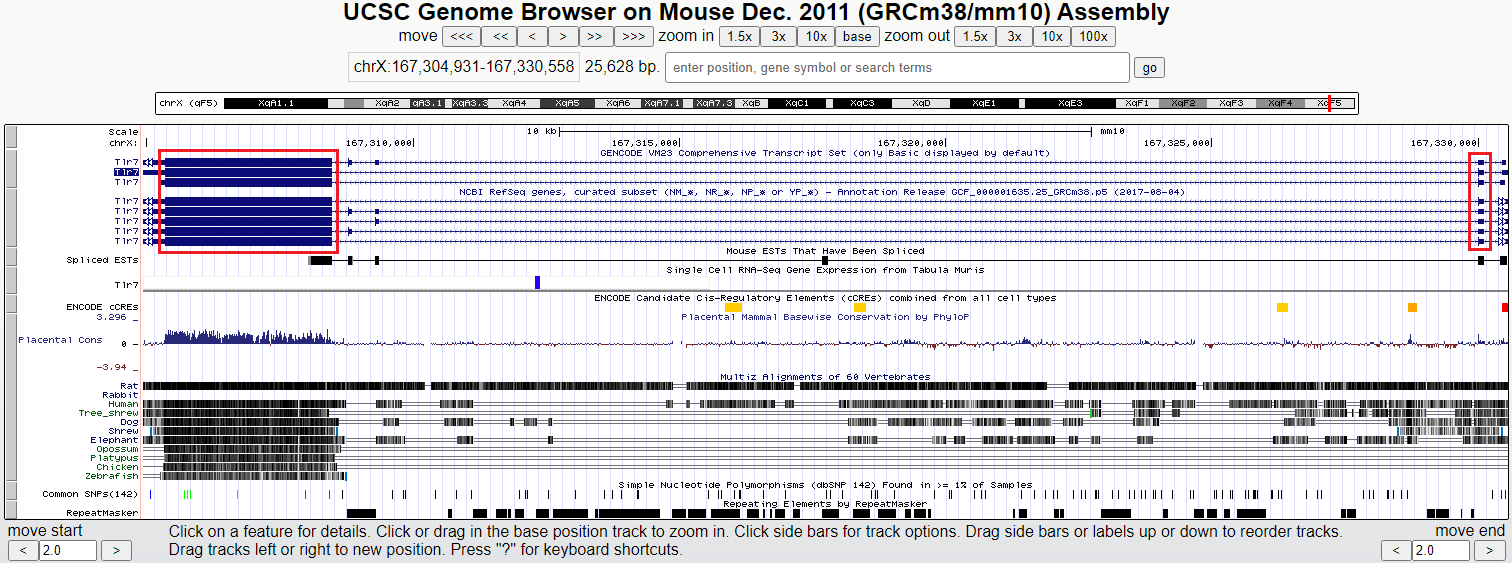
Lúc này, UCSUC Genome Browser sẽ hiện ra tất cả các transcript variant của gene Tlr7. Khi rê chuột vào hình bạn sẽ thấy các vùng exon và intron của gene. Một số transcript variant có 5 exon, trong khi các transcript variant khác chỉ có 3 hoặc 4. Như vậy, chiến lược để cặp mồi có thể nhận ra tất cả các transcript variant là chọn hai exon chung mà variant nào cũng có. Trong hình, hai exon được chọn là hai vùng được đóng khung màu đỏ để minh họa.
Bạn click chuột phải vào tên của một transcript variant và chọn Get DNA sequence for Tlr7. Một trang mới tên là Get DNA for sẽ hiện ra. Trong mục Sequence Format Option, bạn chọn extended case/color option. Trang Extended DNA Case/Color Options hiện ra. Bạn chọn Lower cho Default case và chọn Toggle case cho Spliced ETS đồng thời điền số 255 cho ô Red. Mục đích của việc này là để khi trình tự gene hiện ra, vùng exon sẽ là chữ cái in hoa và có màu đỏ, còn vùng intron sẽ là chữ cái in thường và có màu đen. Bạn cũng có thể chọn mà xanh lá hay xanh dương thay vì màu đỏ tùy theo sở thích của bạn. Sau khi hoàn thành, bạn bấm nút Submit.

Khi chuỗi trình tự DNA hiện ra, bạn cần xác định chuỗi màu đỏ (là vùng exon) tương ứng với các exon nào trong hình 1. Ở đây, chuỗi màu đỏ đầu tiên và chuỗi màu đỏ áp cuối là hai vùng exon mà chúng ta quan tâm (tương ứng với hai exon được đóng khung đỏ trong hình 1).
Bước 2: Thiết kế mồi bằng công cụ PrimerQuest của IDT
IDT là một công ty cung cấp các sản phẩm công nghệ sinh học cho các phòng thí nghiệm. Chúng ta sẽ sử dụng công cụ PrimerQuest của IDT để thiết kế mồi. Địa chỉ của PrimerQuest tại đây: https://www.idtdna.com/Primerquest/Home/Index
Trong mục Sequence Entry: Enter sequence(s) manually, bạn sẽ dán chuỗi trình tự của hai vùng exon mà bạn chọn ở bước 1. Để làm bước này, bạn quay lại trang có chuỗi trình tự DNA ở bước 1. Với chuỗi màu đỏ đầu tiên (exon đầu), bạn copy tầm 100 base cuối cùng của chuỗi. Với chuỗi màu đỏ áp cuối (exon thứ hai), bạn chọn tầm 100 base đầu tiên của chuỗi. Hình 3 minh họa hai khu vực copy như đã miêu tả.

Bạn dán lần lượt hai đoạn copy này vào mục Enter sequence(s) manually của PrimerQuest. Trong mục Choose Your Design, bạn chọn qPCR 2 Primer Intercalating Dyes. Công cụ sẽ hiển thị kết quả. Để chọn cặp mồi phù hợp, bạn lưu ý Start và Stop của mỗi mồi. Exon đầu chúng ta chọn tầm 100 base và exon sau cũng tầm 100 base, do đó start và stop của mỗi mồi phải nằm trong hay khu vực này. Điểm lưu ý thứ hai là GC%. Thường bạn nên chọn cặp mồi có GC% tầm 45-60%. Trong ví dụ này, PrimerQuest cho ra 3 cặp primer khả thi. Bạn bấm vào View Assay Detail để thấy chuỗi trình tự của cặp mồi mà bạn quan tâm. Bài viết sẽ chọn cặp mồi đầu tiên. Bạn cần kiểm tra xem hai mồi có đoạn này tự bắt cặp lẫn nhau hay không.
Bước 3: Kiểm tra cặp mồi bằng in-silico PCR
Để kiểm tra xem cặp mồi có thực sự nhận ra tất cả các transcript variant của gene Tlr7 hay không, chúng ta sẽ dùng công cụ in-silico PCR của UCSC Genome Browser: https://genome.ucsc.edu/cgi-bin/hgPcr
Trong mục Genome, bạn chọn Mouse. Mục Assembly bạn chọn Dec. 2011 (GRCm38/mm10). Mục Target, bạn chọn genome assembly.
Bạn copy cặp mồi mà bạn thu được từ bước 2 vào ô Forward Primer và Reverse Primer. Mục Max product size, bạn điền 100000; sau đó bấm Submit. UCSC In-silico PCR sẽ cho ra kết quả là sản phẩm giả định nếu cặp mồi hoạt động. Bạn bấm vào kết quả để xem trong UCSC Genome Browser.

Dựa trên các kết quả thu được chúng ta tin rằng cặp mồi này có thể nhận ra tất cả các transcript variant. Độ dài của sản phẩm là 98 bp, phù hợp với giới hạn của RT-qPCR (trung bình giới hạn là 400 bp).
Như vậy, chúng ta đã hoàn thành việc thiết kế mồi cho phản ứng RT-qPCR bằng hai công cụ tiện dụng là UCSC Genome Browser và PrimerQuest. IBSG hy vọng rằng bài viết sẽ hữu ích cho các bạn trong việc làm các thí nghiệm liên quan tới PCR.
Huy Vũ